Nat Rev | 如何用数字生物学和AI预测T细胞特异性?
原创 huacishu 图灵基因 2023-04-09 10:11 发表于江苏
收录于合集#前沿生物大数据分析

撰文:huacishu
IF=108.555
推荐度:⭐⭐⭐⭐⭐
亮点:
1、作者阐述了抗原结合预测模型的一般要求,强调了关键的挑战,并讨论了单细胞技术和机器学习等数字生物学的最新进展如何提供可能的解决方案;
2、作者强调了预测TCR特异性如何有助于我们理解抗原免疫原性这一更广泛的谜题。

核心词汇
:TCR(T细胞受体);MHC(主要组织相关性抗原,也就是MHC编码的蛋白质);监督学习(利用一组已知类别的样本调整分类器的参数,使其达到所要求性能的过程);无监督学习(根据类别未知(没有被标记)的训练样本解决模式识别中的各种问题)。
英国牛津大学Hashem Koohy教授课题组在国际知名期刊
Nat Rev Immunol
在线发表题为“
Can we predict T cell specificity with digital biology and machine learning?
”的论文。机器学习和实验生物学的最新进展为长期以来难以解决的蛋白质结构预测等问题提供了突破性的解决方案。然而,尽管T细胞受体(TCR)在协调健康和疾病中的细胞免疫中发挥着关键作用,但从TCR到其同源抗原的可靠图谱的计算重建仍然是系统免疫学的重要领域。

本文中作者提出了新的、协调的跨学科研究,以解决预测TCR抗原特异性的问题。作者阐述了抗原结合预测模型的一般要求,强调了关键的挑战,并讨论了单细胞技术和机器学习等数字生物学的最新进展如何提供可能的解决方案。最后,作者描述了预测TCR特异性如何有助于我们理解抗原免疫原性这一更广泛的谜题。
T细胞通常通过表面表达的高度多样的异二聚体T细胞受体(TCR)识别MHC蛋白家族成员上呈现的抗原(图1)。
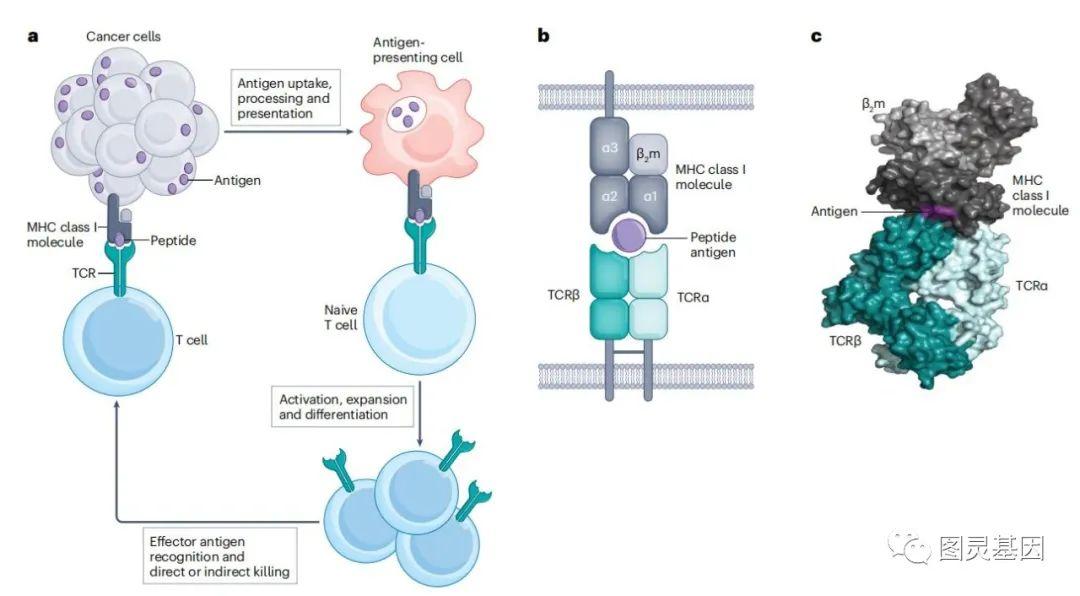
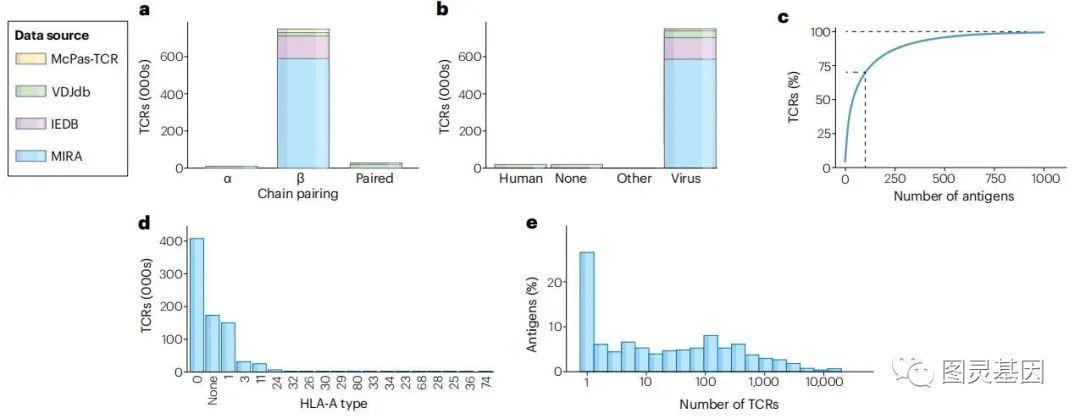
TCR在监测和应对疾病以及开发新疫苗和疗法中有关键的作用。然而,成本和实验问题限制了可用的数据库,这些数据集也不能很好地代表自我和致病性表位以及它们可能出现的各种MHC环境(图2)。
因此,研究界已转向机器学习模型,作为预测TCR的抗原特异性的手段。准确预测TCR抗原特异性可以描述为推导两个相关问题的计算解决方案:首先,给定未知抗原特异性的TCR,最可能结合哪种抗原-MHC复合物;第二,给定抗原MHC复合物,哪些是最可能的同源TCR?
试图回答这些问题的模型的一个关键要求是,它们应该能够对TCR和抗原-MHC复合物的任何组合做出准确的预测。这些应包括模型训练数据中包含的“可见”对和模型未暴露的新的或“不可见”TCR表位对。
我们还必须对预测TCR特异性和抗原免疫原性的相关任务进行重要区分。前者,也是本文的重点,是预测TCR集合与抗原MHC复合物之间的结合。后者可以描述为预测给定抗原是否会诱导功能性T细胞免疫应答。作者认为,只有在细胞和组织层面整合抗原呈递、TCR识别和效应器功能的知识,我们才能充分认识到基础和转化科学的益处。
目前技术水平
实验方法
抗原-MHC多聚体可用于使用大量(合并)T细胞群或更新的单细胞方法来确定TCR特异性。批量的方法被广泛使用且相对便宜,但不提供关于αβ TCR链配对或功能的信息。因此,单链TCR序列在公共数据集中占主导地位(图2)。
多模式单细胞技术提供了对细胞分辨率下的链配对、转录组和表型特征的洞察,但仍然非常昂贵,与批量实验相比,每次运行返回的TCR序列更少,并且显示出对具有高特异性的TCR的显著偏向。减少非特异性多聚体结合的适当实验方案、正确折叠的验证和信噪比的计算改进仍然是有争论领域。
合成肽显示库(synthetic peptide display libraries)的出现(图3a)允许将结合分析扩展到每个TCR数十万肽。这样的高通量文库筛选为改进抗原MHC空间的筛选提供了机会,但限制了对单个TCR的分析。
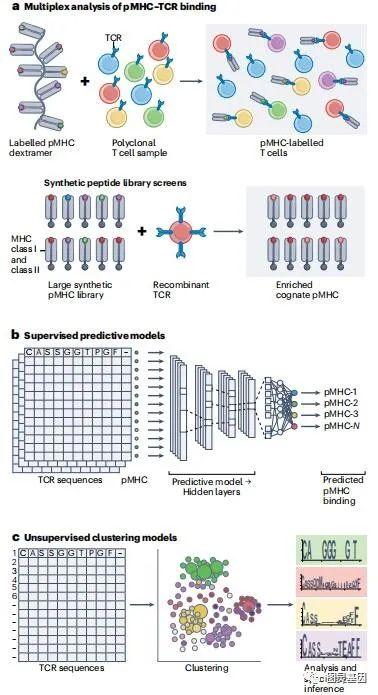
计算方法
广义地说,当前的模型可以分为两类,称之为监督预测模型(SPM)(图3b)和无监督聚类模型(UCM)(图3c),因为它们分别使用了监督学习和无监督学习。
监督预测模型
SPM是将正确预测未知特异性的给定输入TCR的同源表位,给定已知TCR-肽对的一些训练数据集。在过去的两年中,旨在通过深度神经网络(DNN)解决这一挑战的出版物数量有所增加。尽管有许多可能的方法来比较SPM性能,但最常用的方法是ROC曲线下方的面积(ROC-AUC)。
无监督聚类模型
与SPM不同,UCM不依赖于标记数据的可用性,而是学习产生TCR、抗原或HLA输入的分组,这些分组反映了数据的潜在统计变化(图3c)。
主要挑战
尽管未标记的免疫库数据呈指数级增长,并且最近在数据科学和人工智能领域取得了前所未有的突破,但定量免疫学仍然缺乏一个系统推断TCR T细胞抗原特异性的框架。对这些失败的最可信的解释是数据的局限性、方法上的差距和基础免疫学模型的不完整。
结论
总之,数据可用性、方法学和免疫学背景的局限性在机器学习和数字生物学时代的T细胞免疫学领域留下了巨大的空白。作者认为,通过利用来自单细胞数据的大量未标记TCR序列,应用数据增强技术抵消标记数据中的表位和HLA失衡,结合序列和结构感知特征,并应用基于丰富功能和结合数据的前沿计算技术,实现TCR抗原特异性推断的改进。
首先,应提供已标记和未标记TCR数据的合并和验证库,以便于模型预训练和系统比较。其次,应协调努力,提高由不太常见的HLA等位基因和非病毒表位所呈现的TCR抗原对的覆盖率。第三,对SPM、UCM和两者组合的模型性能进行独立、无偏见和系统的评估。未来,TCR特异性推断数据应扩展到包括多模态信息,作为从TCR结合到免疫原性预测的桥梁。
这项任务的规模和复杂性意味着需要一种跨学科的联合方法,以便在组织层面系统地结合细胞免疫的最新免疫学理解以及人工智能和数据科学领域的前沿发展。这应该包括实验和计算免疫学家、机器学习专家以及翻译和工业合作伙伴。考虑到蛋白质结构预测系列的关键评估的成功,鼓励采用类似的方法来解决短期内TCR特异性推断的巨大挑战,并最终预测整合的T和B细胞免疫原性。
教授介绍

Hashem Koohy教授就职于英国牛津大学。2010年,Hashem Koohy教授获得了沃里克大学系统生物学博士学位。接着在剑桥大学桑格研究所做了一项关于转录调控和基因功能的博士后研究。随后,他前往Babraham研究所,使用将多组学融入尖端机器学习技术的小鼠发育B细胞,研究与年龄相关的适应性免疫系统退化相关的表观遗传特征。2017年,他成为牛津大学人类免疫单位(MRC分子医学)的组长。Koohy团队的研究主要集中在病原体的T细胞识别上,其中来自最前沿的大批量和单细胞技术的多模式高通量测序“大数据”被纳入计算、机器学习和统计模型,以研究抗原处理和呈现以及T细胞识别和功能。这也促成了许多国家和国际合作,在许多研究主题中与具有影响力的出版物进行了合作。
参考文献
Hudson D, Fernandes RA, Basham M, Ogg G, Koohy H. Can we predict T cell specificity with digital biology and machine learning?. Nat Rev Immunol. 2023;1-11. doi:10.1038/s41577-023-00835-3
>>>关于我们<<<
2022年热文TOP10
1、
Nat Rev Immu | 线粒体如何控制调节炎症
2、
Science | 靶向蛋白降解途径突破,几乎可用于任何疾病相关蛋白
3、
Cell Metab 丨 靶向肿瘤微环境中CD8+T细胞代谢的治疗潜力
4、
Nature Cancer | 肿瘤相关成纤维细胞的单细胞分析
5、
Annu Rev 丨B细胞在肿瘤微环境中的功能
6、
Nature | 线粒体膜形态如何影响CD4+ T细胞分化
7、
Nat Immun| 肿瘤浸润巨噬细胞的复杂作用
8、
Nat Rev | 巨噬细胞作为癌症治疗的工具和靶点
9、
Science | 癌症中的三级淋巴器官结构
10、
Cancer Cell | 肿瘤免疫新进展:全基因组筛选发现T细胞耗竭调节因子

图灵基因面向科研工作者推出的 TB-1 数字病理切片扫描仪,自带AI建模和模型解释功能,有望帮助您发现更多细胞和组织空间结构规律。
简单易用,轻松构建原创模型,成本可控,门槛低联系微信号:aipathology 电话:18121496883 (刘女士)