【机器学习-因果推断】因果森林 causalForest 估计 HTE R语言官方案例2
前面我们已经介绍过因果树的 HTE 估计 官方案例:
王几行xing:【机器学习-因果推断】因果树 causalTree 估计 HTE R语言快速上手小案例1
估计结果如下图:
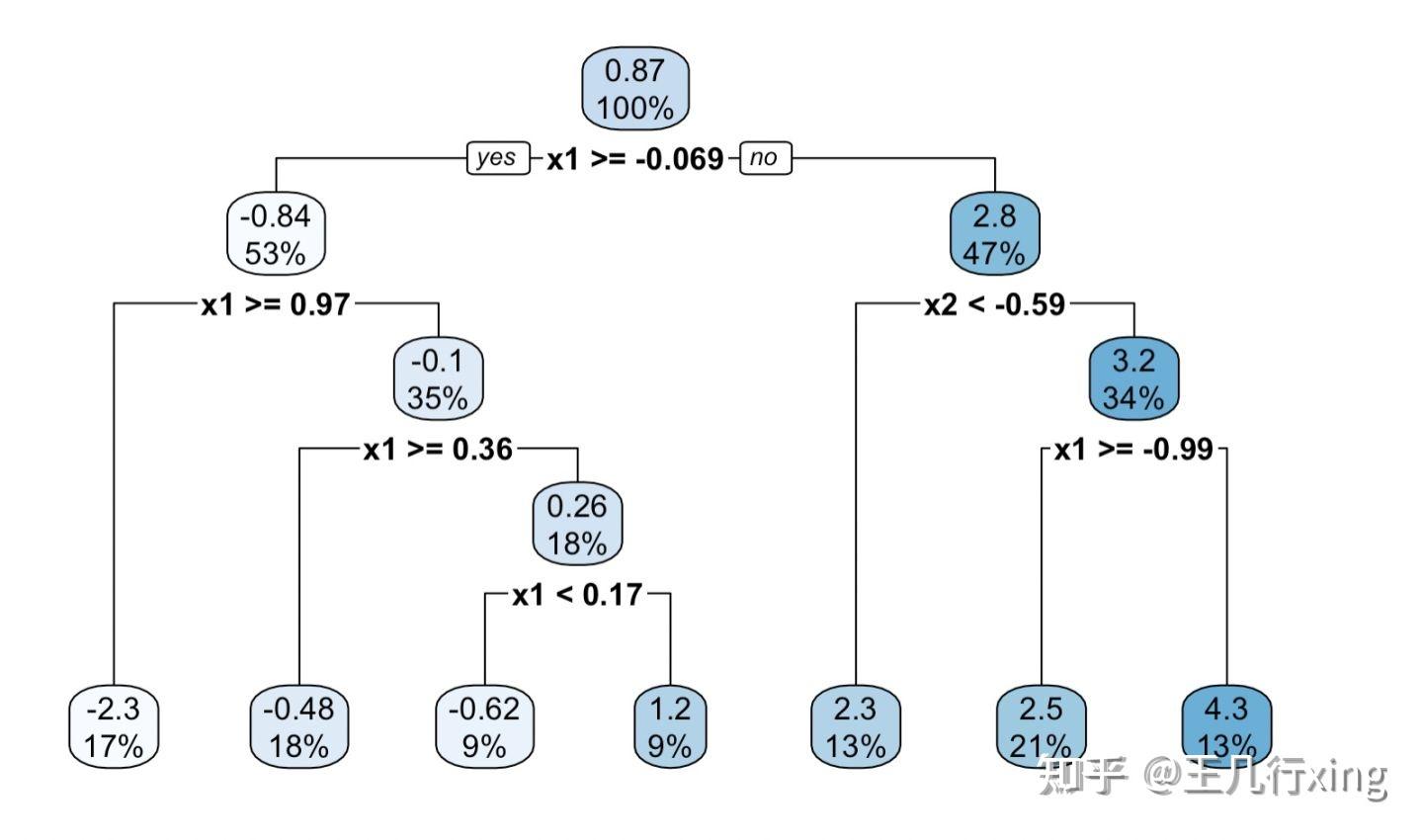
这此基础之上,我们再来介绍因果树的升级--因果森林的官方案例。
1. 环境准备
## RStudio 安装
install.packages("grf")
## Conda 环境安装
conda install -c conda-forge r-grf
## 开发版
devtools::install_github("grf-labs/grf", subdir = "r-package/grf")2. 数据虚构
## 数据虚构
n <- 2000 ## 样本数
p <- 10 ## 列数
## 训练数据
X <- matrix(rnorm(n * p), n, p) ## dim(X) = 2000*10
## 测试数据
X.test <- matrix(0, 101, p) ## dim(X.test) = 101*10
## 修改测试数据的第一列的取值为 -2到2的均匀取值
X.test[, 1] <- seq(-2, 2, length.out = 101)
## 测试数据,虚构干预变量 W 和结果变量 Y
W <- rbinom(n, 1, 0.4 + 0.2 * (X[, 1] > 0)) ## 0-1取值,各一半,共2000行1列
Y <- pmax(X[, 1], 0) * W + X[, 2] + pmin(X[, 3], 0) + rnorm(n) ## 共2000行1列3. 因果森林建模
## 实例化(?)一个因果森林
tau.forest <- causal_forest(X, Y, W) ## 默认树的数量最小值是 2000
## 基于训练数据,袋外方法估计 TE
tau.hat.oob <- predict(tau.forest)
hist(tau.hat.oob$predictions)
4. 基于测试数据,估计 TE
## 基于测试样本, 估计TE
tau.hat <- predict(tau.forest, X.test) ## dim(X.test) = [101, 10]
## 基于 X.test 的第一列 画出 HTE
## X.test[, 1] = seq(-2, 2, length.out = 101)
plot(X.test[, 1], tau.hat$predictions, ylim = range(tau.hat$predictions, 0, 2), ## 黑色实线
xlab = "x", ylab = "tau", type = "l")
lines(X.test[, 1], pmax(0, X.test[, 1]), col = 2, lty = 2) ## 红色虚线
5. 基于全样本,计算CATE和CATT

这里的条件,指的是基于 X 第一个变量的取值。
6. 计算 HTE 的置信区间
## 增加树的数量,计算 HTE 的置信区间
tau.forest <- causal_forest(X, Y, W, num.trees = 4000) ##树的最小数量是 4000
tau.hat <- predict(tau.forest, X.test, estimate.variance = TRUE)
sigma.hat <- sqrt(tau.hat$variance.estimates)
## 画出置信区间
## 黑色实线
plot(X.test[, 1], tau.hat$predictions, ylim = range(tau.hat$predictions + 1.96 * sigma.hat, tau.hat$predictions - 1.96 * sigma.hat, 0, 2), xlab = "x", ylab = "tau", type = "l")
## 上黑色虚线
lines(X.test[, 1], tau.hat$predictions + 1.96 * sigma.hat, col = 1, lty = 2)
## 下黑色虚线
lines(X.test[, 1], tau.hat$predictions - 1.96 * sigma.hat, col = 1, lty = 2)
## 红色实线
lines(X.test[, 1], pmax(0, X.test[, 1]), col = 2, lty = 1)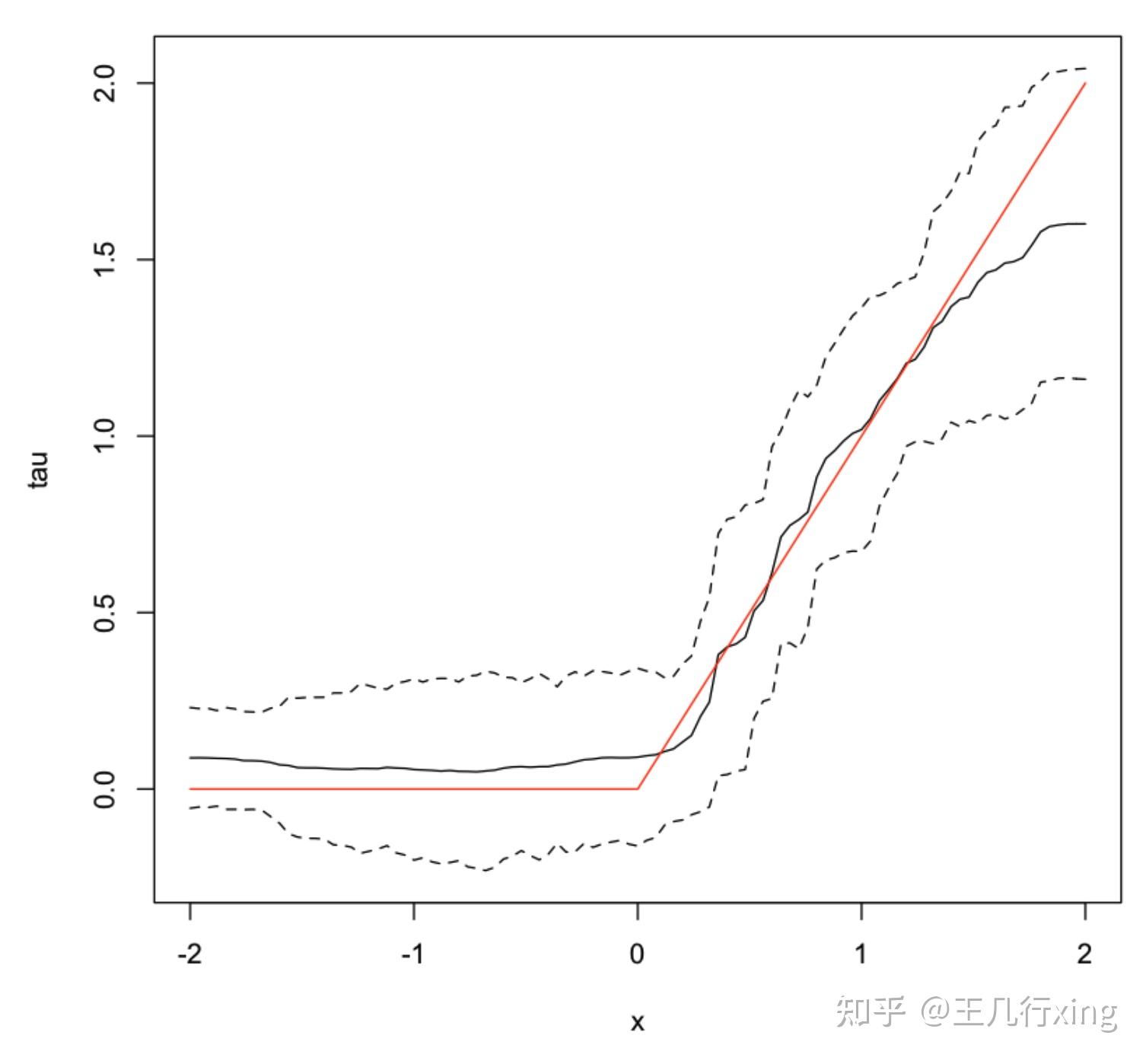
附:用不同方法估算 Yhat 和 What:
## 重新生成数据
n <- 4000
p <- 20
X <- matrix(rnorm(n * p), n, p)
TAU <- 1 / (1 + exp(-X[, 3]))
W <- rbinom(n, 1, 1 / (1 + exp(-X[, 1] - X[, 2])))
Y <- pmax(X[, 2] + X[, 3], 0) + rowMeans(X[, 4:6]) / 2 + W * TAU + rnorm(n)
## 估算 What, 基于回归森林
forest.W <- regression_forest(X, W, tune.parameters = "all")
W.hat <- predict(forest.W)$predictions
## 估算Yhat,基于回归森林
forest.Y <- regression_forest(X, Y, tune.parameters = "all")
Y.hat <- predict(forest.Y)$predictions
forest.Y.varimp <- variable_importance(forest.Y)
## 如果变量过少,那么森林类的算法将会变得不容易实现
## 不应该删除过多的控制变量
selected.vars <- which(forest.Y.varimp / mean(forest.Y.varimp) > 0.2)
## 先查看下控制变量的重要程度
## 如果变量过少,那么森林类的算法将会变得不容易实现
## 不应该删除过多的控制变量
selected.vars <- which(forest.Y.varimp / mean(forest.Y.varimp) > 0.2)
## 重新基于因果森林进行估计
tau.forest <- causal_forest(X[, selected.vars], Y, W,
W.hat = W.hat, Y.hat = Y.hat,
tune.parameters = "all")
## 输出最终结果
test_calibration(tau.forest)
[Out: ]
Best linear fit using forest predictions (on held-out data)
as well as the mean forest prediction as regressors, along
with one-sided heteroskedasticity-robust (HC3) SEs:

