pySCENIC单细胞转录因子分析更新(2):python版分析及可视化
****pySCENIC全部往期精彩系列
:
1、
PySCENIC(一):python版单细胞转录组转录因子分析
2、
PySCENIC(二):pyscenic单细胞转录组转录因子分析
3、
PySCENIC(三):pyscenic单细胞转录因子分析可视化
4、
PySCENIC(四):pyscenic结果之差异转录因子分析及其他可视化
5、
pySCENIC单细胞转录因子分析更新:数据库、软件更新
最近公众号小伙伴好像扎堆做单细胞转录因子富集分析,这里还是建议使用服务器,因为自己电脑可能跑起来比较费劲。我们也在上一篇内容里面对分析进行了更新,我们提供的方法都在liunux终端的conda环境中运行。可以直接运行,也可以在docker镜像中运行。之前我们提供过在pyscenic运行结束后得到loom文件进行分析可视化。这里我们新补充内容,就是python版的可视化。首先解决一个问题,其实这个问题在RNA速率分析的时候我们已经提到了(
单细胞RNA速率分析(3):Python版RNA速率分析分析及可视化
)。我们是在R语言里面提取的seurat单细胞的矩阵去python中分析,所以最后可视化的时候需要将R里面的文件转化为python可读可操作的对象。我们封装了两个函数,第一个是R里面数据提取seurat_to_adata.R,第二个是python中数据构建函数seurat_to_adata.py。接下来就进行可视化分析:
1、TF ranks

2、TF热图
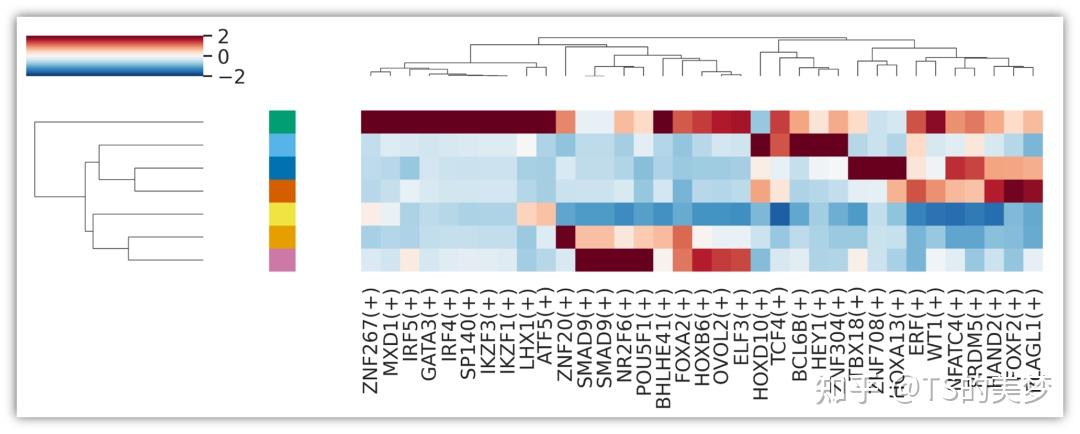

3、转录因子活性聚类图

