FORGE2:GWAS 的细胞类型特异性分析
给大家安利一款软件
FORGE2
,全称
Functional element Overlap analysis of the Results of GWAS Experiments version 2
。简单来说,就是通过对 GWAS 信号位点和基因组功能调控元件进行富集,找到 GWAS 特异的细胞类型。
该工具提供了网页版和命令行版。
一、网页版
网页版 (网址: https:// forge2.altiusinstitute.org / ) 的比较简单,只需要输入RS号即可。
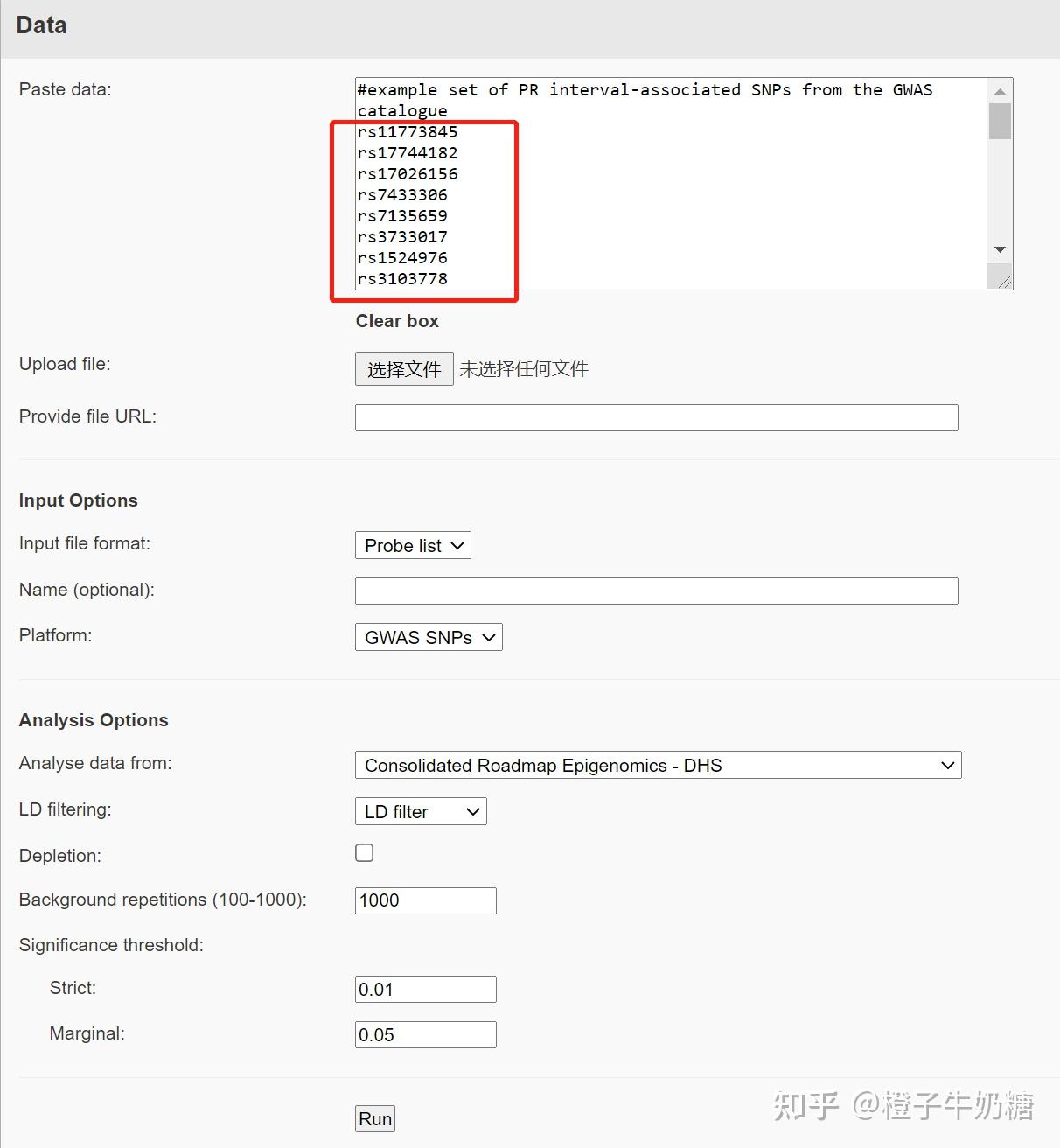
得到以下结果:

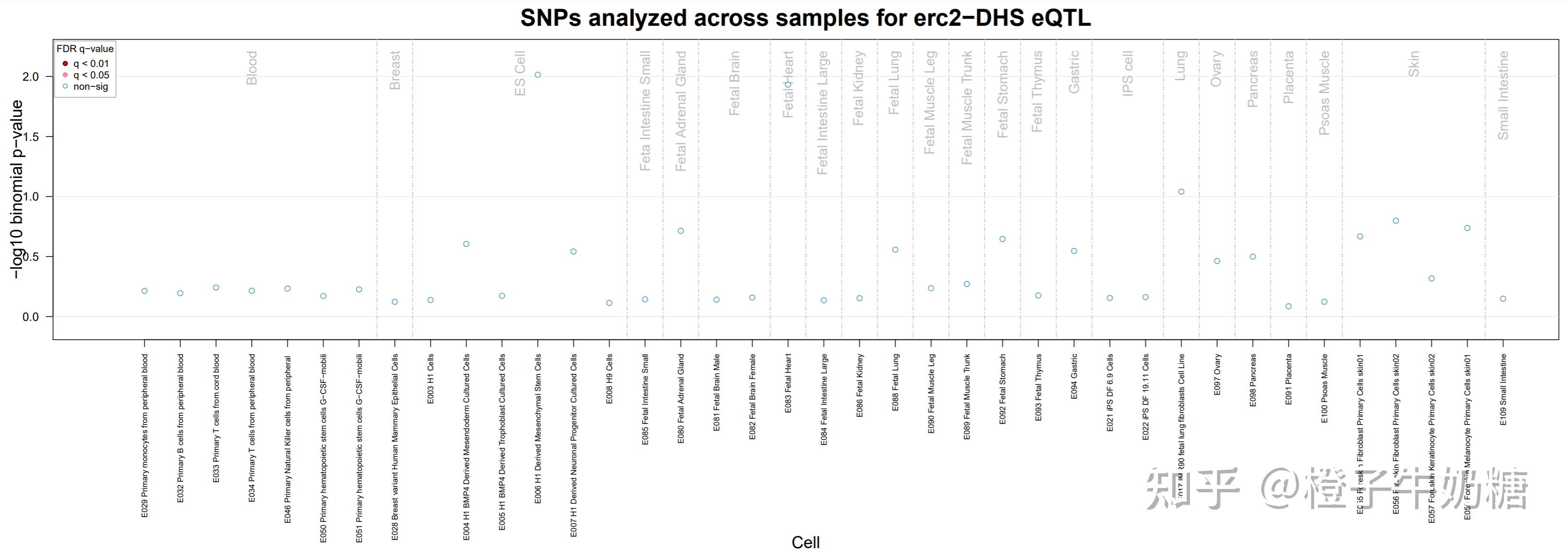
- 以 DHS 的分析为例,会返回以下三张图表:
SNPs analyzed across samples for erc2−DHS
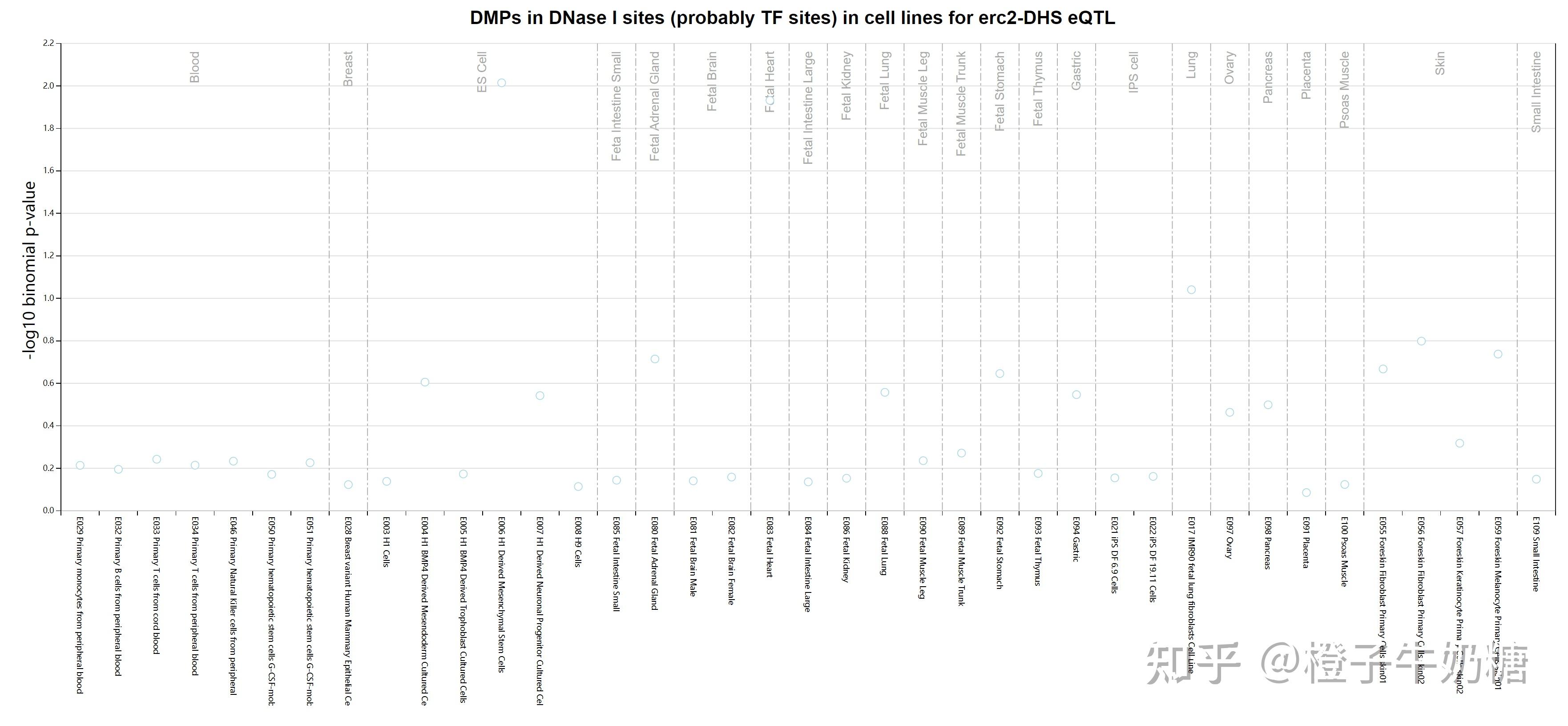
DMPs in DNase I sites (probably TF sites) in cell lines for erc2-DHS
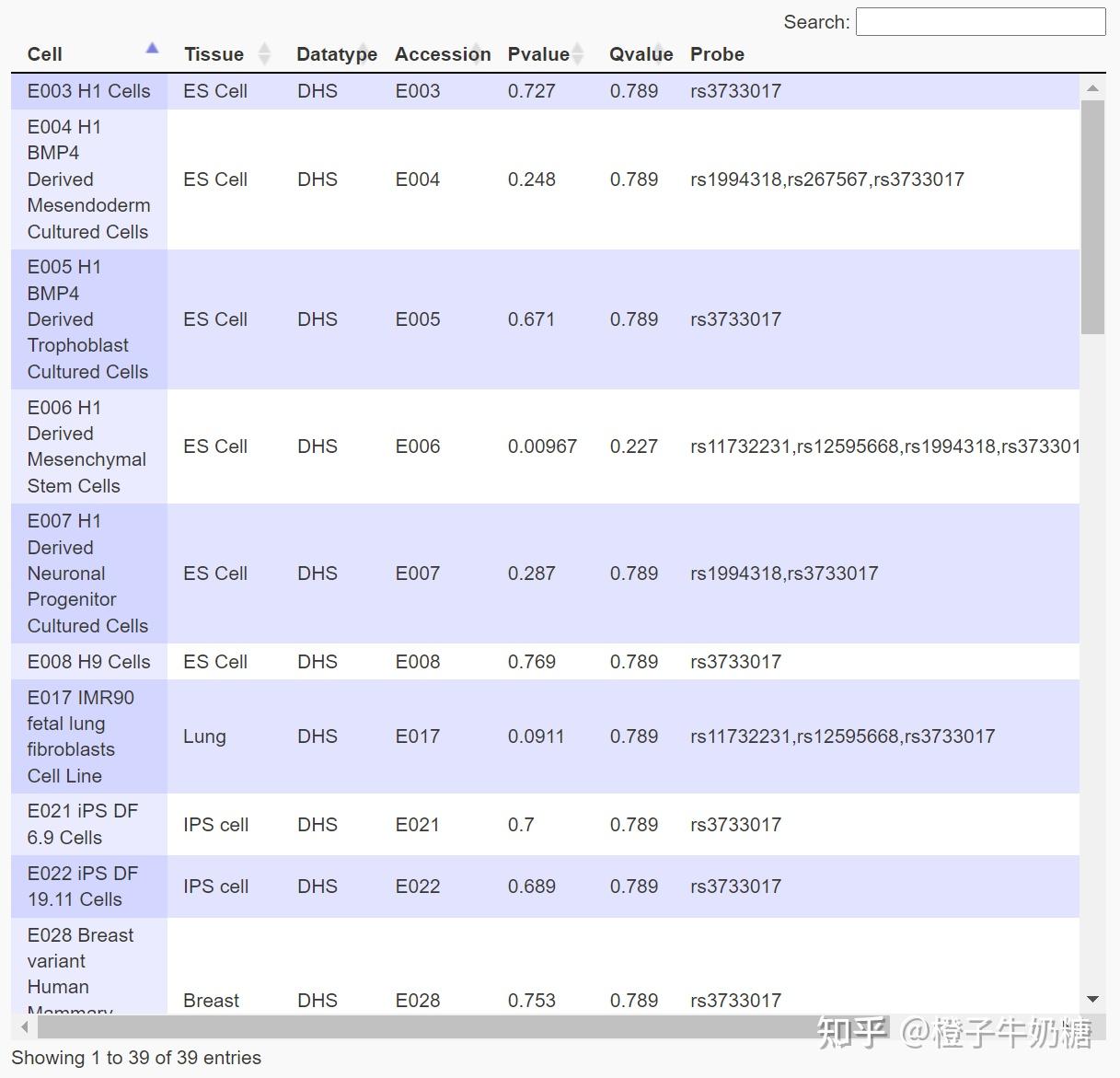
Interactive table
二、命令行版
1、首先下载、解压 FORGE2
wget https://github.com/charlesbreeze/FORGE2/archive/refs/heads/forge2.v1.0.zip
unzip forge2.v1.0.zip2、随后安装 perl 以及 perl 需要的依赖包
conda create -n perl #创建perl环境;
conda activate perl #激活perl环境;
conda install -c conda-forge perl #安装perl;
conda install -c bioconda perl-dbd-sqlite #安装perl模块
conda install -c bioconda perl-sort-naturally #安装perl模块
conda install -c bioconda perl-storable #安装perl模块
conda install -c bioconda perl-getopt-long #安装perl模块
